真菌及与植物互作的表观遗传调控机理研究团组
组长:单淳敏 博士、研究员

研究方向
1.真菌异染色质组装、表观遗传修饰及其跨代遗传的分子调控机制
2.植物-真菌互作的表观遗传学调控机制
研究内容和意义
1. 真菌异染色质组装、表观遗传修饰及其跨代遗传的分子调控机制
真核生物基因组DNA与组蛋白和其他蛋白结合,并折叠形成染色质。组蛋白的共价修饰、染色质重塑、以及DNA修饰等在程序性的基因表达过程中都发挥着重要的调控作用。在相同类型的母代细胞和子代细胞中,基因表达图谱往往是相似甚至一致的。这是因为在母代细胞中,异染色质形态一旦建成后便形成了表观遗传记忆(epigenetic memory)。表观遗传记忆这一重要过程是如何被调控的目前尚不明确。本研究组拟通过各种高通量遗传筛选、分子生物学和生物化学手段,深入发掘并研究表观遗传记忆的调控过程和相关重要的调节因子,解析其调控机制。
2. 植物-真菌互作的表观遗传学调控机制
真菌是植物病害的最主要病原之一,每年给全球的农业生产带来巨大损失。植物真菌病害一般是由病原真菌、环境因子和寄主植物相互作用和影响而形成,因而研究植物与病原真菌的相互作用,以及病原真菌本身的发育调控机制具有重要意义。表观遗传调控在真核生物的生长发育过程中处于核心地位,在植物的免疫反应及病原真菌的侵染过程中也发挥了重要作用。本研究组拟通过各种遗传学、分子生物学和生物化学手段,深入研究植物与病原真菌的相互作用如何通过基因组水平组蛋白甲基化和DNA甲基化的变化,影响下游相关致病基因的表达;同时研究这些异染色质造成的沉默基因簇是否与致病真菌的侵染性有直接联系,并进一步阐明其作用机制。
研究队伍
— 研究组长
单淳敏 研究员、博士生导师
电话:010-64806131
电子邮件:shancm#im.ac.cn(请将#换成@)
通讯地址:北京市朝阳区北辰西路1号院3号中国科学院微生物研究所
邮政编码:100101
— 主要学习及工作经历
2003.09-2007.07 南京大学, 生命科学学院, 生物科学理科基地班, 生物学学士
2007.09-2014.08 中国科学院大学/中科院上海生命科学研究院植物生理生态研究所, 遗传学博士
2014.10-2019.09 美国哥伦比亚大学, 博士后
2019.10-2021.10 美国哥伦比亚大学, 副研究科学家
2021.10-2022.11 中国科学院微生物研究所, 项目研究员
2022.11- 中国科学院微生物研究所, 研究员
— 获奖情况
2021 入选国家海外高层次青年人才引进计划
— 团队

— 工作人员
王海婷 助理研究员
— 在读研究生
杨杰 博士生(2022-)
董慧珍 直博生(2023-)
张超越 博士生(2024-)
刘孟冉 硕士生(2022-)
姜芃 硕士生(2023-)
胡煜笛 硕士生(2024-)
李飔媛 硕士生(2025-)
— 联合培养人员
龙海江 博士生 (广西大学,2024-)
颜昌海 硕士生(山东农业大学,2023-)
莫庭鸣 硕士生(广西大学,2024-)
王湘 硕士生(广西大学,2024-)
— 客座人员
梅世惠 硕士生(贵州大学,2024-)
代表性论文
*, 通迅作者; #, 共同第一作者
2022年及以后:
Toda T#, Fang Y#, Shan CM#*, Xu H, Kim JK, Tang L, Jovanovic M, Tong L, Qiao F, Zhang ZG, Jia S* (2024) Mrc1 regulates parental histone segregation and heterochromatin inheritance. Molecular Cell; 84(17):3223-3236.e4. doi: 10.1016/j.molcel.2024.07.002. (Cover story)
Fang Y, Xu H, Shan CM, Toda T, Qiao F, Zhang Z, Jia S* (2024) Coordination of histone chaperones for parental histone segregation and epigenetic inheritance. Genes and Development; 38: 189-204. (Cover story)
Yang J#, Liu M#, Jiao Y, Guo HS, Shan CM*, Wang H* (2024) An Efficient Homologous Recombination-Based In Situ Protein-Labeling Method in Verticillium dahliae. Biology 13 (2), 81.
Giacomini G, Piquet S, Chevallier O, Dabin J, Bai S, … Shan CM, Reid R, Toda T, … Rondinelli B*, Polo S* (2024) Aberrant DNA repair reveals a vulnerability in histone H3.3-mutant brain tumors. Nucleic Acids Research; 52(5): 2372-2388.
Miao Z#, Wang H#, Tu X#, Huang Z, Huang S, Zhang X, Wang F, Huang Z, Li H, Jiao Y, Gao S, Zhou Z, Shan CM*, Li J*, Yue JX* (2023) GetPrimers: A generalized PCR-based genetic targeting primer designer enabling easy and standardized targeted gene modification across multiple systems. Yeast; https://doi.org/10.1002/yea.3916
Shan CM, Fang Y, Jia S* (2023) Leaving histone unturned for epigenetic inheritance (Invited review). The FEBS Journal; doi:10.1111/febs.16260
Du W#, Shi G#, Shan CM#, Li Z#, Zhu B*, Jia S*, Li Q*, Zhang ZG* (2022) Mechanisms of chromatin-based epigenetic inheritance (Invited Review). Science China Life Sciences, 65(11):2162-2190.
2021年及以前:
Shan CM, Kim JK, Wang J, Bao K, Sun Y, Chen H, Yue JX, Stripe A, Schalch T, Liti G, Nagy PL, Tong L, Qiao F, Jia S* (2021) The histone H3K9M mutation synergizes with H3K14 ubiquitylation to selectively sequester histone H3K9 methyltransferase Clr4 at heterochromatin. Cell Reports; 35(7):109137.
Shan CM, Bao K, Diedrich J, Chen X, Chao Lu, Yates JR, Jia S* (2020) The INO80 complex regulates epigenetic inheritance of heterochromatin. Cell Reports; 33(13):108561.
Bao KH#, Shan CM#, Moresco JJ, Yates JR, Jia S*. Antisilencing factor Epe1 recruits SAGA to promote transcription within heterochromatin (2018) Genes and Development; 33(1-2): 116–126.
Zhang Y#, Shan CM#, Wang J#, Bao K, Tong L*, Jia S*. (2017) Molecular basis for the role of oncogenic histone mutations in modulating H3K36 methylation. Scientific Reports 7, 43906; doi: 10.1038/srep43906.
Shan CM#, Wang J#, Xu K#, Chen H, Yue JX, Andrews S, Moresco JJ, Yates JR, Nagy PL, Tong L*, Jia S*. (2016) A histone H3K9M mutation traps histone methyltransferase Clr4 to prevent heterochromatin spreading. eLife; doi: 10.7554/eLife.17903
Shan CM, Shangguan XX, Zhao B, Zhang XF, Chao L, Yang CQ, Wang LJ, Zhu HY, Zen YD, Guo WZ, Zhou BL, Hu GJ, Guan XY, Chen ZJ, Wendel JF, Zhang TZ*, Chen XY.* (2014) Control of cotton fiber elongation by a homeodomain transcription factor, GhHOX3. Nature Communications; 5:5519; doi: 10.1038/ncomms6519.
Bao K, Shan CM, Chen X, et al. (2022) The cAMP signaling pathway regulates Epe1 protein levels and heterochromatin assembly. PLoS Genetics 18(2): e1010049
Cao JF#, Zhao B#, Huang CC, Chen ZW, Zhao T, Liu HR, Hu GJ, Shangguan XX, Shan CM, Wang LJ, Zhang TZ, Wendel JF, Guan XY, Chen XY* (2020) The miR319-targeted GhTCP4 promotes the transition from cell elongation to wall thickening in cotton fiber. Molecular Plant 13 (7), 1063-1077
Liu X#, Zhao B#, Zheng HJ#, Hu Y#, Lu G, … Shan CM, Jiang JP, Zhu YQ, ... Zhang GY*, Wang SY, Zhang TZ*, Zhao GP, Chen XY* (2015) Gossypium barbadense genome sequence provides insight into the evolution of extra-long staple fiber and specialized metabolites. Scientific Reports 5:14139;
Yu ZX, Wang LJ, Zhao B, Shan CM, Zhang YH, Chen DF, and Chen XY*. (2015) Progressive regulation of sesquiterpene biosynthesis in Arabidopsis inflorescence by the miR156-Targeted SPL transcription factors. Molecular Plant 8 (1), 98-110
Yu N#, Cai WJ#, Wang SC, Shan CM, Wang LJ, Chen XY* (2010) Temporal control of trichome distribution by microRNA156-targeted SPL Genes in Arabidopsis thaliana. The Plant Cell 22: 2322–2335.
Guan XY, Li QJ, Shan CM, Wang S, Mao YB, Wang LJ, Chen XY* (2008) The HD-Zip IV gene GaHOX1 from cotton fiber is a functional homologue of the Arabidopsis GLABRA2 gene. Physiologia Plantarum. 134: 174–182
代表性图片
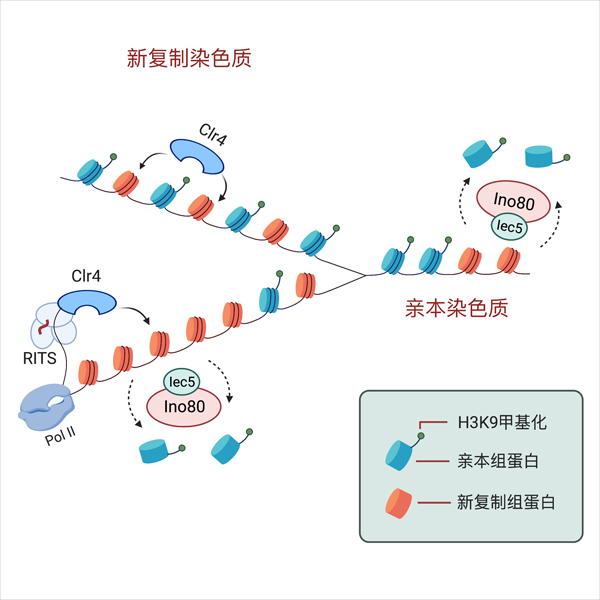
组蛋白甲基化表观记忆遗传的受染色质重塑复合体调控
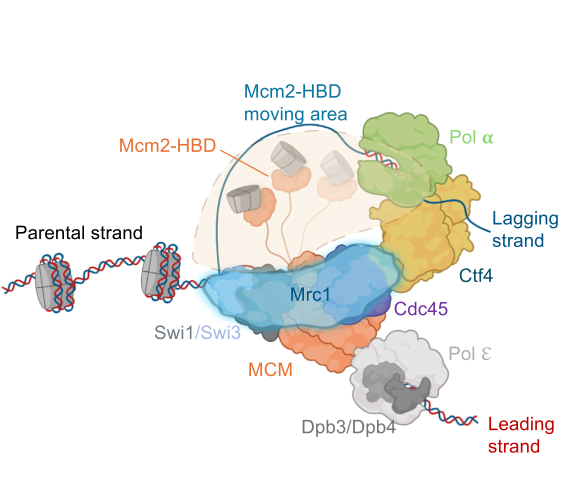
组蛋白甲基化表观记忆遗传的调控机制
申请专利
1. 单淳敏; 王海婷; 谭永浚琳; 杨杰 ; 一种高效检测真菌全基因组水平组蛋白修饰位点和丰度的方法, 2024-02-08, 中国, 202410177158X
2. 单淳敏; 王海婷; 谭永浚琳; 杨杰 ; 一种高效检测真菌全基因组水平DNA结合蛋白的分布与丰度的方 法, 2024-02-08, 中国, 202410177000.2
3. 陈晓亚; 赵波; 单淳敏; 上官小霞; 杨长青; 王凌健 ; GhPRE1基因及其在改良棉纤维品质中的应用, 2016-03-03, 中国, CN201610121078.8
4. 陈晓亚; 单淳敏; 关雪莹; 上官小霞; 王水; 王凌健 ; HOX3基因在改良棉纤维伸长性状中的用途, 2015-6-24, 中国, CN201510355077.5
5. 陈晓亚; 徐冰; 林芝萍; 王凌健; 上官小霞; 单淳敏 ; 提高植物产量的基因工程方法及材料, 2012- 12-12, 中国, CN201210094113.3
参考示例:
http://www.im.cas.cn/jgsz2018/yjtx/zgkxybywswymyxzdsys/201811/t20181109_5168622.html




