DNA组装与DNA合成技术并称为合成生物学的两大基础使能技术。在合成生物学和生物工程领域中,“设计-构建-验证-学习(Design-Build-Test-Learn,DBTL)”的循环工程,精细的遗传元件无缝拼接以及重复DNA序列(如CRISPR和TALEN结合序列)串联分别需要迭代、无痕和序列重复的DNA组装技术。目前酶切连接的组装方法被广泛的应用于迭代组装,其中BioBrick组装法则是最早发展起来的DNA标准化组装技术,已成为国际基因工程机器大赛iGEM的组装标准。然而,BioBrick及其类似的组装方法在DNA连接处形成6–21 bp的疤痕序列,影响DNA完整性和mRNA正确折叠,且不适用于序列严格依赖的遗传元件的精确组装。
中科院微生物所病原室温廷益研究组建立了基于IIP和IIS型限制性内切酶(Restriction Endonuclease,RE)的DNA组装方法(命名为PS-Brick)。与在两端加入RE识别序列的现有DNA组装技术不同,本方法仅在PS-Brick供体DNA的一端巧妙设计了毗邻的IIP和IIS型RE识别序列,并且保留了另外一端的PCR产物特性,解决了目前基于RE的DNA组装方法普遍存在“疤痕”序列问题。该技术使用IIP型RE切割供体DNA产生一个粘性末端,同时保留PCR产物的另一个平末端或加A粘性末端;使用IIP和IIS型RE同时切割载体,IIP型RE产生粘性末端,IIS型RE产生平末端或单碱基粘性末端,双酶切后IIS型RE的识别序列脱离载体。由相同IIP型RE切割的供体和载体的粘性末端进行连接,组装后的载体重新产生与初始载体相同的毗邻IIP/IIS型RE识别序列对,作为下一轮PS-Brick组装的入口,从而实现迭代组装。供体DNA的平末端或加A粘性末端与IIS型RE切割的载体平末端或单碱基粘性末端进行连接,形成无“疤痕”序列的连接“焊点“,从而实现无痕组装。从供体DNA制备到组装产物转化感受态细胞并涂布平板,一轮PS-Brick组装可在一个工作日内完成。PS-Brick方法的组装效率达到104–105 CFUs/μg DNA,组装正确率约为90%,满足常规分子克隆和建库的需求。
本研究进一步应用PS-Brick方法开展了苏氨酸和正丙醇代谢工程的研究。应用PS-Brick迭代组装的特性,开展了多轮 DBTL循环的代谢工程,逐步解除了关键酶ThrA的反馈抑制、消除了代谢瓶颈、增强了外排转运途径以及阻断了降解途径,构建了高效的苏氨酸和正丙醇工程菌。除了迭代组装的优点,PS-Brick无痕组装实现了ThrA饱和突变以及调控翻译起始的双顺反子元件的精确拼接;PS-Brick重复序列组装实现了3个含有相同启动子和sgRNA的CRISPR基因组编辑序列的串联整合。
本研究建立了简单高效的PS-Brick组装方法,可同时实现迭代、无痕和序列重复的DNA组装。现有的DNA组装技术难以同时实现这三种组装性能,因此PS-Brick为合成生物学和代谢工程提供了一种有价值的使能技术。该研究已于近日发表于Biotechnology for Biofuels,微生物所刘树文博士和博士生肖海涵为该文的共同第一作者,温廷益研究员为通讯作者。该研究得到中国科学院战略性先导专项A(鸿鹄专项,XDA17010503)、国家自然科学基金(31800073)和中科院青促会(2018117)等项目的资助。
文章链接:https://biotechnologyforbiofuels.biomedcentral.com/articles/10.1186/s13068-019-1520-x
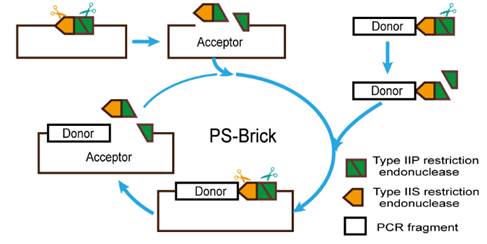
PS-Brick组装流程图




